近日,Advanced Science(IF=15.1)在线发表了江南大学生物工程学院周哲敏教授团队的研究成果“A New-Generation Base Editor with An Expanded Editing Window for Microbial Cell Evolution In Vivo Based on CRISPR‒Cas12b Engineering”(Hao et al., Adv. Sci., 2024, DOI: 10.1002/advs.202309767)。2019级博士生郝文亮(已毕业,现为清华大学化学工程系2023级博士后)为论文的第一作者,周哲敏教授和韩来闯助理研究员为论文的共同通讯作者。
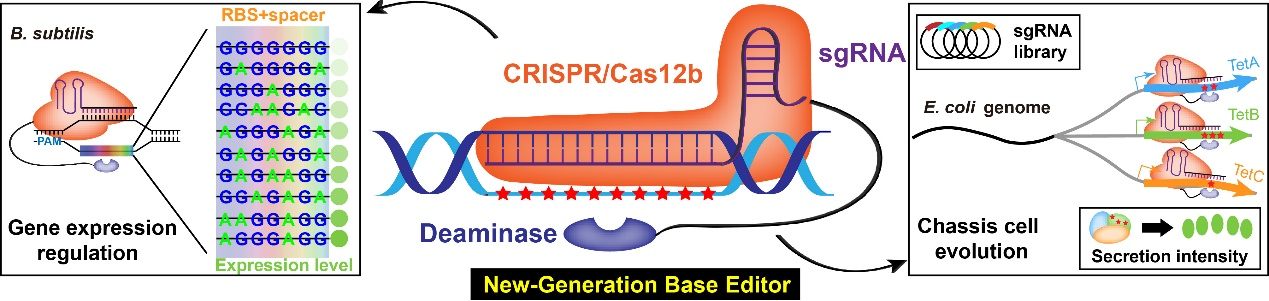
图1 新一代瘦身版碱基编辑器开发摘要
碱基编辑器(Base editor,BE)作为一种新兴的靶向突变技术已经被广泛用于微生物细胞进化、蛋白质工程、代谢工程以及合成生物学的其他方面研究。对于微生物细胞进化而言,往往需要在一个较大的编辑窗口内,通过产生大量突变以提高表型的多样性。然而,微生物细胞中现存的BE编辑窗口比较狭窄(仅5~7 nt),至多发生2~3个氨基酸的替换,限制了BE的应用。尽管通过sgRNA的延长以及多类型脱氨酶的融合可以在一定程度上扩展BE的编辑窗口,但是其在靶标处的可编辑范围依然有限。
针对上述关键科学问题,本研究提出使用分子量较小的Cas12b构建BE系统,其与脱氨酶融合可以尽量减弱Cas蛋白的大空间位阻对于脱氨酶编辑窗口的限制效应。首先在重要模式工业菌株枯草芽孢杆菌中构建了CRISPR/BhCas12b系统,结合多序列比对及BhCas12b/sgRNA复合体结构预测模型,确定BhCas12b上负责DNA切割的三个关键残基(D574,D828和D952),并通过失活突变构建了完全丧失DNA切割能力、但保留与sgRNA高效结合及定位靶标基因能力的dBhCas12b。

图2 序列-结构导向的dBhCas12b构建
将胞嘧啶脱氨酶CDA融合到dBhCas12b的N端并额外添加两个拷贝的UGI(CDA-dBhCas12b-UGI-UGI)成功构建CBE体系,可以在16 nt的编辑窗口诱发C到T的突变。另外,将改造后的腺苷脱氨酶ABE融合到dBhCas12b的N端构建ABE体系,可以在14 nt的编辑窗口高效诱发的A到G突变。新一代CBE和ABE编辑窗口分别是目前报道的2~5倍。
将CBE系统用于枯草芽孢杆菌目标基因RBS+Spacer的大窗口(15 nt)共进化,构建RS组合突变文库,筛选得到eGFP表达强度提升68.1倍的最佳RS组合突变。另外,在E. coli中也测试了本CBE系统的宿主适用性,结果发现,该CBE在E. coli也同样适用,能够在PAM两端(以PAM为0点)同时诱导C到T的突变(C-15~C26),编辑窗口高达41 nt,甚至超出了protospacer(23 nt)。全基因组测序证明,dBhCas12b-CBE并没有发生可检测的脱靶效应,证明了该系统是一个高精度的基因组编辑器。

图3 RBS+Spacer的原位进化提高基因表达水平
利用新一代BE编辑窗口的优势,设计构建了靶向E. coli Tat分泌系统的sgRNA文库用于开发高分泌性能的E. coli底盘。经过基因组编辑以及荧光筛选实验分析,获得了一株蛋白分泌性能提升6.49倍的E. coli底盘,证明了该CBE系统在微生物底盘宿主进化中的应用潜力。测序结果显示,在E. coli宿主中新一代BE的编辑窗口是目前已报道BE的8~10倍,是迄今微生物系统中编辑窗口最宽的BE。
本项目得到国家重点研发计划(2023YFC3402400)等项目的资助。

图4 高分泌性能E. coli底盘开发
