近期,我校生物工程学院邓禹教授课题组在大肠杆菌核心启动子设计与强度调控方面取得重要进展,研究成果“Deep learning guided programmable design of Escherichia coli core promoters from sequence architecture to strength control”正式发表于Nucleic Acids Research(IF = 13.1)。
精准预测和理性设计细菌核心启动子一直是合成生物学领域的重大挑战。核心启动子是调控转录起始的关键元件,其序列空间高度紧凑,即使极微小的碱基变动都可能带来显著的功能差异。由于其序列与功能关系高度非线性,并伴随复杂的上下文依赖,研究者难以依靠经验规则或有限实验全面解析其中规律。与此同时,现有启动子数据集覆盖范围有限、分辨率不足,缺乏对大规模变异序列的系统性测量,进一步制约了高精度模型的建立。
近年来,人工智能方法逐渐应用于转录调控研究,但多数依赖于边界模糊的基因组序列,难以严格界定功能区段;或依赖于转录组学水平的间接信号(如RNA表达量),而这些并不能真实反映启动子的转录活性。由此导致预测结果与实际功能之间常常存在偏差,难以支撑高精度的理性设计,尚不能满足合成生物学对可编程基因表达调控的迫切需求。
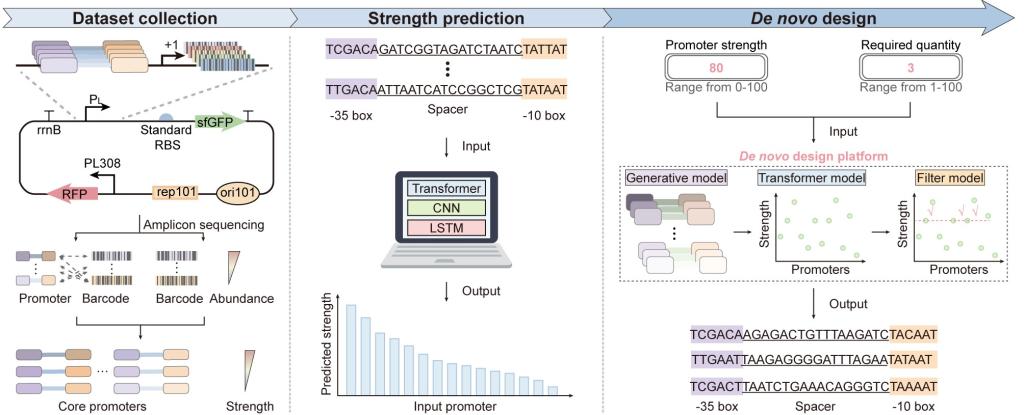
针对上述问题,江南大学生物工程学院邓禹教授课题组开发了一个集理性文库构建、预测建模与生成式设计于一体的综合平台,实现对大肠杆菌核心启动子的可编程调控。团队创新性地提出并应用MBRS(Mutation-Barcoding-Reverse Sequencing)策略,构建了一个包含112,955个启动子的高质量数据集,覆盖16,226倍表达范围。在此基础上训练的Transformer模型不仅实现了高精度预测(相关系数 R = 0.87),还揭示了与经典基序及上下文依赖特征相吻合的注意力模式。进一步结合条件扩散模型,该平台能够从头生成具有精确目标强度的新型启动子(R = 0.95),并在不同遗传背景中展现出良好的泛化能力(R = 0.93)。设计出的启动子在组成型与可诱导系统中均表现出稳定的模块化“即插即用”调控效果。该平台已免费开放,访问地址为www.yudenglab.com。
论文通讯作者为邓禹教授、周胜虎副研究员和刘洋副教授,我校2021级博士研究生周璇为第一作者。此研究工作得到了国家重点研发计划(2024YFA0918000)、江苏省杰出青年科学基金(BK20220089)、江苏省重点研发计划(现代农业)(BE2022322)、国家自然科学基金(22378170和22478156)和物联网“试点计划”专项(2022SP-T16-B)等资助。
近年来,邓禹教授团队深耕代谢工程、合成生物学与系统生物学领域,围绕 基因表达调控元件的设计与应用展开系统研究。团队在可编程启动子、RBS设计以及动态调控网络构建等方面取得了系列创新成果,有效推动了微生物细胞工厂的构建与优化。相关研究成果已发表在Nucleic Acids Research (2025、2020)、Advanced Science (2025、2024)、Chemical Engineering Journal (2025)、Bioresource Technology (2024)、ACS Catalysis (2023)、ACS Sustainable Chemistry & Engineering (2023)、Metabolic Engineering (2022、2021)等本领域权威期刊,部分研究成果已在企业中得到推广应用。
