近期,我校生物工程学院倪晔教授团队在机器学习辅助酶立体选择性的定向进化方面取得重要进展,研究成果“Enhanced stereodivergent evolution of carboxylesterase for efficient kinetic resolution of near-symmetric esters through machine learning”正式发表于Nature Communications (IF = 14.7) (https://doi.org/10.1038/s41467-024-53191-8)。
手性环己-3-烯-1-羧酸(CHCA)具有一个几乎对称的六原子环,是合成各种药物、农用化学品和天然产物的关键组成部分。这些底物通常被认为是“难以区分”的,被化学催化剂和生物催化剂都难以识别。定向进化在加速立体选择性酶的开发中起着关键作用,但在催化“难以区分”的底物时,也面临一定的挑战。机器学习(ML)作为一种强大的计算工具,为基于大量高质量数据和统计模型促进定向进化提供了更直接的捷径。
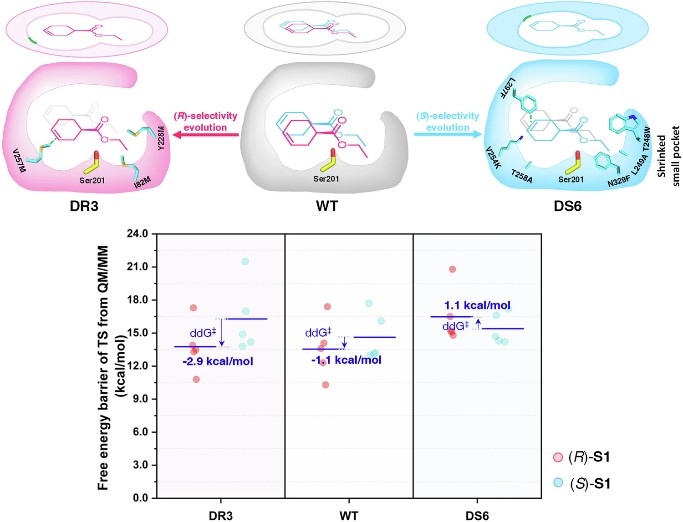
研究团队首先开发了一种高通量方法来生成羧酸酯酶AcEst1的高质量数据集,利用羧酯酶的生化特征包括体积、疏水性、亲水性、静电、氢键、π-π相互作用和到催化残基的距离用于训练ML模型,评估并训练了包括核岭回归(KRR)、高斯过程回归(GPR)、梯度提升回归树(GBRT)、随机森林回归(RFR)、支持向量回归(SVR)和贝叶斯岭回归(BRR)6个回归模型在AcEst1的对映选择性与7个生化特征之间的相关性。将经过训练的ML预测模型应用于设计组合突变体,成功实现了羧酸酯酶AcEst1立体选择性的发散进化,构建了(R)-选择性和(S)-选择性的突变酶DR3和DS6,并在1 M底物浓度下实现(R)和(S)-CHCE的合成与分离,并采用分子动力学(MD)模拟及QM/MM计算解释了羧酯酶及其突变体立体选择性调控的分子机制。
我校毕业生2019级博士窦哲为第一作者,许国超副教授和倪晔教授为论文的共同通讯作者。上述研究得到了国家重点研发计划(2019YFA0906401)、国家自然科学基金(22078127、22378169)等项目资助。
近年来倪晔教授团队通过酶工程以及生物催化等技术,在生物法高效合成高值化合物的方面取得了丰硕成果,相关研究成果已发表在Journal of the American Chemical Society、ACS Catalysis、Chemical Engineering Journal、ACS Sustainable Chemistry & Engineering、ACS Synthetic Biology等本领域权威期刊。
